新智元报道
编辑:静音
六个月的争议后,诺奖级 AI AlphaFold3 开源了。这个在蛋白质结构预测领域掀起波澜的 AI——期待它的开源推动更多科学家的大量创新。文后附有安装和运行步骤详解哦!
AlphaFold3 源码终于开放了!
六个月前,AlphaFold3 横空出世震撼了整个学术界。AlphaFold 的开发人也凭借它在上个月赢得了诺贝尔化学奖。

然而,这个诺奖级 AI 的「不开源」一直引起学界的不满。谷歌 DeepMind 只推出了一个免费研究平台「AlphaFold Server」,而且该服务有每日的次数限制。相比于开源的 AlphaFold2 来说,这种使用方式缺失了很多自由度。
好在它现在终于开源了!开源后,生化医药的科学家们可以在本地部署 AlphaFold3,极大地缩短了新药、疫苗等研发进程。
现在,任何人都可以下载 AlphaFold3 软件代码并进行非商业使用,但目前只有学术背景的科学家可申请访问训练权重。
GitHub 上的 AlphaFold3 开源项目代码目前已斩获 1.8k 星。
开源项目:https://github.com/google-deepmind/alphafold3
AlphaFold3 的「效仿者」们
在过去的几个月中,不少公司都依靠 AlphaFold3 论文中的伪代码,争相发布了各自受到 AlphaFold3 启发的类似模型。
比如,获得 OpenAI 投资的 AI 生物初创 Chai Discovery,就在 9 月发布了用于分子结构预测的新型多模态基础模型 Chai-1,并附带了一份技术报告,比较了 Chai-1 与 AlphaFold 等模型的性能。
官网地址:https://www.chaidiscovery.com/
另一家位于美国旧金山的公司 Ligo Biosciences 则发布了一个无使用限制的 AlphaFold3 版本。但它尚未具备完整的功能,比如模拟药物和蛋白质以外分子的能力。
项目地址:https://github.com/Ligo-Biosciences/AlphaFold3
其他团队也正在开发没有使用限制的 AlphaFold3 版本:AlQuraishi 希望在年底前推出一个名为 OpenFold3 的完全开源模型。这将使制药公司能够使用专有数据(例如结合不同药物的蛋白质结构)重新训练模型,从而有可能提高性能。
开源的重要性
过去一年里,许多公司发布了新的生物 AI 模型,这些公司对开放性采取了不同的态度。
威斯康星大学麦迪逊分校的计算生物学家 Anthony Gitter 对盈利性公司加入他的领域没有异议——只要他们在期刊和预印本服务器上分享工作时遵循科学界的标准。
「我和其他人希望盈利性公司们也分享关于如何进行预测的信息,并以我们可以审查的方式发布 AI 模型和代码,」Gitter 补充道,「我的团队不会基于无法审查的工具进行构建和使用。」
DeepMind 科学 AI 负责人 Pushmeet Kohli 表示,几种 AlphaFold3 复制品的出现表明,即使没有开源代码,该模型也是可复现的。
他补充说,未来他希望看到更多关于出版规范的讨论,因为这一领域越来越多地由学术界和企业研究人员共同参与。
此前,AlphaFold2 的开源推动了其他科学家的大量创新。
例如,最近一次蛋白质设计竞赛的获胜者使用该 AI 工具设计出能够结合癌症靶标的新蛋白质。
AlphaFold 项目的负责人 Jumper 最喜欢的一个 AlphaFold2 创新,是一个团队使用该工具识别出一种帮助精子附着在卵细胞上的关键蛋白。
Jumper 迫不及待地想看到在分享 AlphaFold3 后出现这样的惊喜。
安装和运行
安装 AlphaFold3 需要一台运行 Linux 的机器;AlphaFold3 不支持其他操作系统。
完整安装需要多达 1TB 的磁盘空间来存储基因数据库(建议使用 SSD 存储)以及一块具有计算能力 8.0 或更高的 NVIDIA GPU(具有更多内存的 GPU 可以预测更大的蛋白质结构)。
经过验证,单个 NVIDIA A100 80 GB 或 NVIDIA H100 80 GB 可以适配最多 5120 个 token 的输入。在 NVIDIA A100 和 H100 GPU 上的数值准确性也已被验证。
尤其是对于较长的目标,基因搜索阶段可能会消耗大量 RAM——建议至少使用 64GB 的 RAM 运行。
配置步骤:
1. 在 GCP 上配置机器
2. 安装 Docker
3. 为 A100 安装 NVIDIA 驱动程序
4. 获取基因数据库
5. 获取模型参数
6. 构建 AlphaFold3 Docker 容器或 Singularity 镜像
获取 AlphaFold3 源代码
通过 git 下载 AlphaFold3 的代码库:
git clone https://github.com/google-deepmind/alphafold3.git获取基因数据库
此步骤需要「curl」和「zstd」。
AlphaFold3 需要多个基因(序列)蛋白质和 RNA 数据库来运行:
- BFD small
- MGnify
- PDB(mmCIF 格式的结构)
- PDB seqres
- UniProt
- UniRef90
- NT
- RFam
- RNACentral
Python 程序「fetch_databases.py」可以用来下载和设置所有这些数据库。
建议在「screen」或「tmux」会话中运行以下命令,因为下载和解压数据库需要一些时间。完整数据库的总下载大小约为 252GB,解压后的总大小为 630GB。
cd alphafold3 # Navigate to the directory with cloned AlphaFold3 repository.python3 fetch_databases.py --download_destination=该脚本从托管在 GCS 上的镜像下载数据库,所有版本与 AlphaFold3 论文中使用的相同。
脚本完成后,应该有以下目录结构:
pdb_2022_09_28_mmcif_files.tar # ~200k PDB mmCIF files in this tar.bfd-first_non_consensus_sequences.fastamgy_clusters_2022_05.fant_rna_2023_02_23_clust_seq_id_90_cov_80_rep_seq.fastapdb_seqres_2022_09_28.fastarfam_14_9_clust_seq_id_90_cov_80_rep_seq.fastarnacentral_active_seq_id_90_cov_80_linclust.fastauniprot_all_2021_04.fauniref90_2022_05.fa获取模型参数
访问 AlphaFold3 模型参数需要向 Google DeepMind 申请并获得授权。
数据管线
数据管线的运行时间(即基因序列搜索和模板搜索)可能会因输入的大小、找到的同源序列数量以及可用的硬件(磁盘速度尤其会影响基因搜索的速度)而显著变化。
如果想提高性能,建议提高磁盘速度(例如通过利用基于 RAM 的文件系统),或增加可用的 CPU 核心并增加并行处理。
此外,请注意,对于具有深度 MSA 的序列,Jackhmmer 或 Nhmmer 可能需要超出推荐的 64 GB RAM 的大量内存。
模型推理
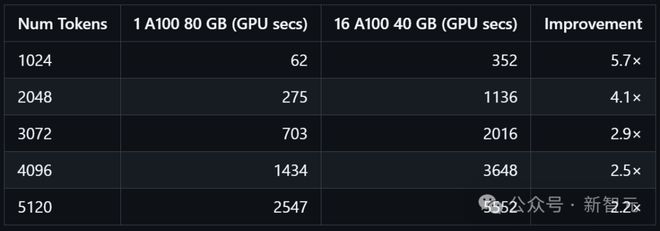
AlphaFold3 论文的补充信息中的表 8 提供了在配置为运行在 16 个 NVIDIA A100 上时的 AlphaFold3 的无需编译的推理时间,每个设备具有 40GB 的内存。
相比之下,该存储库支持在单个 NVIDIA A100 上运行 AlphaFold3,具有 80GB 内存,并在配置上进行了优化以最大化吞吐量。
下表中使用 GPU 秒(即使用 16 个 A100 时乘以 16)比较了这两种设置的无需编译的推理时间。该存储库中的设置在所有 token 大小上效率更高(提高至少 2 倍),表明其适合高吞吐量应用。
硬件要求
AlphaFold3 正式支持以下配置,并已对其进行了广泛的数值准确性和吞吐量效率测试:
- 1 NVIDIA A100(80GB)
- 1 NVIDIA H100(80GB)
通过以下配置更改,AlphaFold3 可以在单个 NVIDIA A100 (40GB) 上运行:
1. 启用统一内存。
2. 调整 model_config.py 中的 pair_transition_shard_spec:
pair_transition_shard_spec: Sequence[_Shape2DType] = ((2048, None),(3072, 1024),(None, 512),)虽然数值上准确,但由于可用内存较少,因此与 NVIDIA A100 (80GB) 的设置相比,该配置的吞吐量会较低。
虽然也可以在单个 NVIDIA V100 上使用 run_alphafold.py 中的--flash_attention_implementation=xla 来运行长度最多为 1280 token 的 AlphaFold3,但此配置尚未经过数值准确性或吞吐量效率的测试,因此请谨慎操作。
参考资料:






